Функционалды геномика - Functional genomics
Функционалды геномика өрісі болып табылады молекулалық биология сипаттауға тырысады ген (және ақуыз ) функциялар және өзара әрекеттесу. Функционалды геномика көптеген деректерді пайдаланады геномдық және транскриптомдық жобалар (мысалы геномдарды ретке келтіру жобалары және РНҚ секвенциясы ). Функционалды геномика ген сияқты динамикалық аспектілерге баса назар аударады транскрипция, аударма, ген экспрессиясының реттелуі және ақуыз-ақуыздың өзара әрекеттесуі сияқты геномдық ақпараттың статикалық аспектілеріне қарсы ДНҚ тізбегі немесе құрылымдар. Функционалды геномиканы зерттеудің негізгі сипаттамасы - олардың осы сұрақтарға жалпы геномды көзқарасы, әдетте дәстүрлі «ген-ген» тәсілінен гөрі жоғары өнімді әдістерді қамтиды.

Функционалды геномиканың анықтамасы және мақсаттары
Функционалды геномиканы түсіну үшін алдымен функцияны анықтау маңызды. Олардың қағазында[1] Граур және басқалар. функцияны екі мүмкін жолмен анықтаңыз. Бұл «Таңдалған әсер» және «Себепті рөл». «Таңдалған әсер» функциясы таңдалатын функцияны білдіреді (ДНҚ, РНҚ, ақуыз және т.б.). «Себепті рөл» функциясы белгінің жеткілікті және қажетті функциясын білдіреді. Функционалды геномика әдетте функциялардың «Себепті рөлі» анықтамасын тексереді.
Функционалды геномиканың мақсаты гендердің немесе ақуыздардың, ақыр соңында геномның барлық компоненттерінің қызметін түсіну. Функционалды геномика термині көбіне көп сілтеме жасау үшін қолданылады техникалық тәсілдер ағзаны зерттеу гендер мен белоктар, соның ішінде «әрбір гендік өнімнің биохимиялық, жасушалық және / немесе физиологиялық қасиеттері»[2] ал кейбір авторларға зерттеуді қосады ноненикалық элементтер олардың анықтамасында.[3] Функционалды геномикаға табиғи зерттеулер де кіруі мүмкін генетикалық вариация біршама уақыттан кейін (мысалы, организмнің дамуы) немесе ғарыш (оның денесінің аймақтары сияқты), сондай-ақ мутациялар сияқты функционалдық бұзылулар.
Функционалды геномиканың уәдесі - организмнің динамикалық қасиеттерін түсіну үшін геномдық және протеомдық білімді қалыптастыру және синтездеу. Бұл геномның функцияны жеке гендерді зерттеумен салыстырғанда қалай анықтайтыны туралы толық көрініс бере алады. Функционалды геномика деректерін интеграциялау көбінесе оның бөлігі болып табылады жүйелік биология тәсілдер.
Техника және қолдану
Функционалды геномикаға геномның функцияға қатысты аспектілері жатады мутация және полиморфизм (сияқты жалғыз нуклеотидті полиморфизм (SNP) талдау), сонымен қатар молекулалық белсенділікті өлшеу. Соңғысына бірқатар «-omics « сияқты транскриптомика (ген экспрессиясы ), протеомика (ақуыз өндірісі ), және метаболомика. Функционалды геномика негізінен қолданады мультиплекс сияқты көптеген гендік өнімдердің көптігін өлшейтін әдістер мРНҚ немесе белоктар ішінде биологиялық үлгі. Функционалды геномикаға бағытталған әдіс бір геннің барлық нұсқаларының қызметін тексеріп, белсенділікті оқудың реті бойынша реттілікті қолдану арқылы мутанттардың әсерін анықтай алады. Бұл өлшеу тәсілдері бірге әр түрлі биологиялық процестердің мөлшерін анықтауға және ген мен ақуыздың қызметі мен өзара әрекеттесуі туралы түсінігімізді жақсартуға тырысады.
ДНҚ деңгейінде
Генетикалық өзара әрекеттесу картасы
Гендерді жүйелі түрде жұппен жою немесе гендердің экспрессиясының тежелуін физикалық өзара әрекеттесуге қатыспаса да, функциясы бар гендерді анықтау үшін қолдануға болады. Эпистаз екі түрлі генді нокаутқа әсер етудің қосымша болмауы мүмкін екендігіне сілтеме жасайды; яғни екі ген тежелгенде пайда болатын фенотип бір нокаут әсерінің жиынтығынан өзгеше болуы мүмкін.
ДНҚ / ақуыздардың өзара әрекеттесуі
Гендердің экспрессиясын реттеуде мРНҚ-ны (ақуыз синтезіне арналған ДНҚ-дан алынған ақпарат, РНҚ, хабарлама) аудару нәтижесінде пайда болған ақуыздар үлкен рөл атқарады. Гендердің экспрессиясын қалай реттейтінін түсіну үшін олардың өзара әрекеттесетін ДНҚ тізбектерін анықтау қажет. ДНҚ-ақуыздың өзара әрекеттесу орнын анықтау әдістемесі жасалды. Оларға жатады Чиптер тізбегі, CUT & RUN реттілігі және қоңырау карталары.[4]
ДНҚ-ға қол жетімділік талдаулары
Геномның қол жетімді аймақтарын анықтау үшін талдаулар жасалды. Ашық хроматиннің бұл аймақтары үміткерлердің реттеуші аймақтары болып табылады. Бұл талдау құрамына кіреді ATAC-сек, DNase-Seq және FAIRE-Seq.
РНҚ деңгейінде
Микроаралдар
Микроариалдар берілген генге немесе зондтың ДНҚ тізбегіне сәйкес келетін үлгідегі мРНҚ мөлшерін өлшейді. Зондтар тізбегі қатты бетке иммобилизденеді және рұқсат етіледі будандастыру флуоресцентті белгіленген «мақсатты» мРНҚ бар. Дақтың флуоресценциясының қарқындылығы сол нүктеге будандастырылған мақсатты дәйектіліктің мөлшеріне, демек, сол мРНҚ тізбегінің көптігіне пропорционалды. Микроариалдар белгілі бір процестерге қатысатын кандидаттардың гендерін идентификациялауға мүмкіндік береді, олар әр түрлі жағдайлардағы транскрипт деңгейлерінің өзгеруіне және белгілі функциялардың гендерімен ортақ экспрессиялық үлгілерге негізделген.
SAGE
Гендердің экспрессиясын сериялық талдау (SAGE) - будандастыруға емес, РНҚ тізбектелуіне негізделген баламалы талдау әдісі. SAGE әр генге ғана тән 10-17 негізгі жұп белгілерінің реттілігіне сүйенеді. Бұл тегтер өндірілген поли-А мРНҚ және тізбектелу алдында ұшынан ұшына дейін байланған. SAGE бір ұяшыққа транскрипттердің санын бейтарап өлшейді, өйткені бұл транскриптерді зерттеудің алдын-ала білуіне тәуелді емес (микроаралар сияқты).
РНҚ секвенциясы
РНҚ секвенциясы соңғы жылдары 2016 жылы атап өткендей, микроарра мен SAGE технологиясын қабылдады және транскрипция мен ген экспрессиясын зерттеудің ең тиімді әдісі болды. Мұны әдетте жасайды келесі буынның реттілігі.[5]
Секвенирленген РНҚ-лардың кіші бөлігі - бұл кішігірім РНҚ, кодталмаған РНҚ молекулаларының класы, олар транскрипциялық және транскрипциядан кейінгі гендердің тынышталуының негізгі реттеушілері немесе РНҚ тынышталуы. Келесі ұрпақтың дәйектілігі - бұл алтын стандартты құрал кодталмаған РНҚ ашу, профильдеу және экспрессияға талдау.
Репортерлердің жаппай параллельдік талдаулары (MPRAs)
Репортерлердің жаппай параллельдік талдауы - бұл ДНҚ тізбектерінің цис-реттеуші белсенділігін тексеру технологиясы.[6][7] MPRA-да синтетикалық генді басқаратын промотордың жоғарғы жағында синтетикалық цис-реттеуші элементі бар плазмида қолданылады, мысалы, жасыл флуоресцентті ақуыз. Сис-реттеуші элементтердің кітапханасы әдетте MPRA көмегімен тексеріледі, кітапханада жүзден мыңға дейін цис-реттеуші элементтер болуы мүмкін. Элементтердің сис-реттеуші белсенділігі төменгі репортерлік қызметті қолдану арқылы талданады. Кітапхананың барлық мүшелерінің қызметі параллельді түрде әрбір cis-реттеуші элемент үшін штрих-кодтар көмегімен бағаланады. MPRA-дің бір шектеуі - белсенділік плазмидада талданады және геномда байқалатын гендердің реттелуінің барлық аспектілерін қамтымауы мүмкін.
STARR-сек
STARR-seq - бұл кездейсоқ қырқылған геномдық фрагменттердің күшейткіш белсенділігін талдау үшін MPRA-ға ұқсас әдіс. Бастапқыда,[8] Drosophila геномының кездейсоқ қырқылған фрагменттері минималды промотордың төменгі жағына орналастырылды. Кандидат күшейткіштері кездейсоқ қырқылған фрагменттердің арасында минималды промоутер көмегімен транскрипция жасайды. Осы әдіспен секвенирлеуді оқылым ретінде пайдалану және әр тізбектің кіріс мөлшерін бақылау арқылы күшейтілген күшейткіштердің күші бағаланады.
Perturb-seq

Perturb-seq жұптары CRISPR генді экспрессиялаумен геннің нокдаунына негізделген. Сызықтық модельдер бір геннің нокдаунының көптеген гендердің экспрессиясына әсерін есептеу үшін қолданылады.
Ақуыз деңгейінде
Ашытқы екі гибридті жүйе
Ашытқы екі гибридті скрининг (Y2H) физикалық ақуыз бен ақуыздың өзара әрекеттесуін анықтау үшін «жем» ақуызын көптеген потенциалды өзара әрекеттесетін белоктарға («жем») қарсы сынайды. Бұл жүйе транскрипция коэффициентіне негізделген, бастапқыда GAL4,[9] ақуыз репортер генінің транскрипциясын тудыруы үшін ДНҚ-мен байланыстыратын және транскрипцияны активтендірудің жеке домендері қажет. Y2H экранында «жем» ақуызы GAL4 байланыстыру аймағына қосылады, ал потенциалды «олжа» (өзара әрекеттесетін) ақуыздардың кітапханасы активация доменімен векторда рекомбинантты түрде өрнектеледі. In vivo ашытқы жасушасындағы жемдер мен жемдер белоктарының өзара әрекеттесуі GAL4 активтенуі мен байланысатын домендерін бір-біріне жеткілікті түрде жақындатады, нәтижесінде репортер ген. Сондай-ақ, жасушадағы барлық өзара әрекеттесулерді анықтау үшін жем ақуыздарының кітапханасын жыртқыш белоктар кітапханасына жүйелі түрде тексеруге болады.
AP / MS
Жақындықты тазарту және масс-спектрометрия (AP / MS) бір-бірімен кешендерде өзара әрекеттесетін ақуыздарды анықтай алады. Белгілі бір «жем» ақуызының айналасында белоктар кешендерінің түзілуіне жол беріледі. Қармақ ақуызы антидене немесе рекомбинантты тег көмегімен анықталады, ол оны өзімен бірге комплекс түзген кез келген ақуыздармен бірге алуға мүмкіндік береді. Содан кейін белоктар қысқа етіп қорытылады пептид фрагменттер мен масс-спектрометрия ақуыздарды сол фрагменттердің зарядталу коэффициенттері негізінде анықтау үшін қолданылады.
Терең мутациялық сканерлеу
Терең мутациялық сканерлеу кезінде алдымен белоктағы аминқышқылдарының кез-келген өзгеруі синтезделеді. Осы ақуыз нұсқаларының әрқайсысының белсенділігі әр нұсқа үшін штрих-кодтар көмегімен параллельді түрде талданады. Белсенділікті жабайы типтегі ақуызбен салыстыру арқылы әр мутацияның әсері анықталады. Комбинаторикаға байланысты аминқышқылдардың кез-келген мүмкін өзгеруін талдау мүмкін болғанымен, екі немесе одан да көп қатарлас мутацияны сынап көру қиын. Терең мутациялық сканерлеу тәжірибелері ақуыздың құрылымын және ақуыз-ақуыздың өзара әрекеттесуін анықтау үшін де қолданылды.
Функцияны жоғалту әдістері
Мутагенез
Гендердің жұмысын гендерді жүйелі түрде «нокаутпен» бір-бірлеп зерттеуге болады. Мұны екеуі де орындайды жою немесе функцияны бұзу (мысалы инерционды мутагенез ) және пайда болған организмдер бұзылған геннің функциясын анықтайтын фенотиптерге тексеріледі *
RNAi
РНҚ-интерференциясы (RNAi) әдістерін синтетикалық ~ 20-мер қысқа интерференцияланған РНҚ молекулаларын (сиРНҚ) трансфекциялау арқылы жеткізілетін ~ 20 негізді жұп екі тізбекті РНҚ-ны пайдаланып уақытша тыныштандыру немесе ген экспрессиясын құлату үшін қолдануға болады. -хайрпиндік РНҚ (шРНҚ). RNAi экрандары, әдетте жасуша дақылына негізделген талдауларда немесе эксперименталды организмдерде орындалады (мысалы C. elegans) геномдағы барлық гендерді немесе гендердің кіші топтарын (суб-геномдар) жүйелі түрде бұзу үшін қолданыла алады; бұзылған гендердің мүмкін функцияларын бақылаулар негізінде тағайындауға болады фенотиптер.
CRISPR экрандары
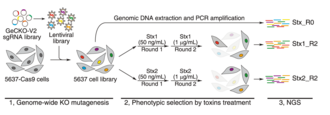
CRISPR-Cas9 гендерді мультиплекстелген жолмен жасуша сызықтарында жою үшін қолданылған. Экспериментке дейін және одан кейін әр ген үшін гид-РНҚ мөлшерін анықтау маңызды гендерге бағытталуы мүмкін. Егер бағыттаушы-РНҚ маңызды генді бұзса, онда бұл жасушаның жоғалуына әкеледі, демек экраннан кейін сол гид-РНҚ-ның сарқылуы болады. Жақында жүргізілген CRISPR-cas9 экспериментінде сүтқоректілердің жасушалық сызықтарында 2000-ға жуық гендер бірнеше жасушалар қатарында маңызды болып табылды.[11][12] Осы гендердің кейбіреулері тек бір жасуша-сызықта қажет болды. Гендердің көп бөлігі көп белокты кешендердің бөлігі болып табылады. Бұл тәсілді тиісті генетикалық фонды қолдану арқылы синтетикалық өлімді анықтау үшін қолдануға болады. CRISPRi және CRISPRa функциялардың жоғалуы және функцияның жоғарылауы экрандарын ұқсас түрде қосады. CRISPRi K562 жасуша желісінде ~ 2100 маңызды генді анықтады.[13][14] CRISPR жою экрандары геннің потенциалды реттеуші элементтерін анықтау үшін де қолданылған. Мысалы, ScanDel деп аталатын әдістеме жарық көрді, ол осы тәсілге тырысты. Авторлар осы геннің реттеуші элементтерін анықтау мақсатында қызығушылық генінен тыс аймақтарды жойды (мендельдік бұзылысқа қатысқан HPRT1).[15] Гассперини және басқалар. осы әдісті қолдана отырып, HPRT1 үшін дистальды реттеуші элементтерді анықтаған жоқ, бірақ мұндай тәсілдерді басқа қызығушылық тудыратын гендерге таратуға болады.
Гендерге арналған функционалды аннотация
Геномдық аннотация
Гипотикалық гендерді геномды белоктарды кодтайтын аймақтарға сканерлеу арқылы анықтауға болады, мысалы, ұзаққа созылатын сипаттамаларға сүйене отырып ашық оқу шеңберлері, транскрипциялық инициация тізбектері және полиаденилдеу сайттар. Болжалды ген ретінде анықталған дәйектілік сол организмнен алынған cDNA немесе EST тізбектеріне ұқсастық, болжамды ақуыздар тізбегінің белгілі белоктарға ұқсастығы, промоторлық тізбектермен байланысы немесе дәйектіліктің мутациясы нәтижесінде пайда болатын дәлелдермен расталуы керек. бақыланатын фенотип.
Розетта тасқа жақындау
Розетта тас тәсілі - бұл де-ново ақуызының қызметін болжау үшін есептеу әдісі. Ол белгілі бір физиологиялық процеске қатысатын кейбір ақуыздар бір организмде екі бөлек ген ретінде, ал екіншісінде бір ген ретінде өмір сүруі мүмкін деген гипотезаға негізделген. Геномдар бір организмде, ал екіншісінде бір ашық оқу шеңберінде тәуелді емес тізбектерге сканерленеді. Егер екі ген бірігіп кеткен болса, онда олардың осындай биорганизмдері ұқсас ко-регуляцияны тиімді ететін ұқсас функциялары болады деп болжануда.
Функционалды геномикаға арналған биоинформатика әдістері
Осы техникамен жасалған мәліметтердің көптігі мен биологиялық мағыналы заңдылықтарды табуға деген ұмтылысынан, биоинформатика функционалдық геномика деректерін талдау үшін өте маңызды. Осы сыныптағы техниканың мысалдары деректер кластері немесе негізгі компоненттерді талдау бақылаусыз машиналық оқыту (сыныпты анықтау), сондай-ақ жасанды нейрондық желілер немесе векторлық машиналар басқарылатын машиналық оқытуға (сынып болжамын, жіктеу ). Функционалды байыту талдауы фондық жиынтықтарға қатысты функционалдық категориялардың артық немесе жеткіліксіз экспрессиясының (РНа экрандарындағы оң немесе теріс реттегіштердің) дәрежесін анықтау үшін қолданылады. Гендік онтология негізделген байыту талдауы қамтамасыз етілген ДӘУІТ және гендер жиынтығын байытуды талдау (GSEA),[16] Тапқырлыққа негізделген талдау [17] және Pathway студиясы[18] және COMPLEAT арқылы ақуызды кешенді талдау.[19]

Терең мутациялық сканерлеу экспериментінің нәтижелерін түсіну үшін жаңа есептеу әдістері жасалды. 'Фидмдер' терең мутациялық сканерлеу экспериментінің нәтижесін филогенетикалық ағашпен салыстырады.[20] Бұл пайдаланушыға табиғаттағы іріктеу процесі ақуызға ұқсас шектеулерді қолданса, терең мутациялық сканерлеу нәтижелері көрсеткендей қорытынды жасауға мүмкіндік береді. Бұл экспериментаторға табиғатты қаншалықты жақсы көрсететіндігіне қарай әр түрлі эксперименттік жағдайларды таңдауға мүмкіндік беруі мүмкін. Терең мутациялық сканерлеу ақуыз бен ақуыздың өзара әрекеттесуін анықтау үшін де қолданылған.[21] Авторлар димердің әртүрлі бөліктеріндегі мутациялардың әсерін болжау үшін термодинамикалық модельді қолданды. Ақуыздың құрылымын шығару үшін терең мутациялық құрылымды да қолдануға болады. Терең мутациялық сканерлеу кезінде екі мутация арасындағы күшті оң эпистаз 3-кеңістіктегі ақуыздың бір-біріне жақын екі бөлігін көрсете алады. Содан кейін бұл ақпаратты ақуыздың құрылымын шығару үшін пайдалануға болады. Бұл тәсіл принципінің дәлелі ретінде GB1 ақуызын қолданатын екі топ көрсетті.[22][23]
MPRA эксперименттерінің нәтижелері деректерді түсіндіру үшін машиналық оқыту тәсілдерін қажет етеді. Үлкен белсенділігі жоқ цис-реттеуші тізбектерде байытылған кмерлерді шығару үшін бос белсенділігі бар SVM моделі пайдаланылды.[24] Бұл модельдер жоғары болжамды күш береді. Осы жоғары өлшемді эксперименттердің нәтижелерін түсіндіру үшін терең оқыту және кездейсоқ орман тәсілдері қолданылды.[25] Бұл модельдер гендік реттеуге қатысты кодталмаған ДНҚ функциясы туралы жақсы түсінік қалыптастыруға көмектеседі.
Функционалды геномикаға бағытталған консорциум жобалары
ENCODE жобасы
ENCODE (ДНҚ элементтерінің энциклопедиясы) жобасы - бұл геномдық ДНҚ-ның барлық функционалды элементтерін кодтау және кодтау аймақтарында анықтау болып табылатын адамның геномын терең талдау. Маңызды нәтижелерге геномдық плиткалық массивтерден көптеген нуклеотидтердің кодтау транскрипциясы, кодталмаған РНҚ немесе кездейсоқ транскрипт ретінде транскрипцияланатындығы, қосымша транскрипциялық регуляторлық орындардың ашылуы, хроматинді өзгертетін механизмдерді одан әрі түсіндіру туралы дәлелдер кіреді.
Генотип-тіндік өрнек (GTEx) жобасы

GTEx жобасы - бұл генетикалық вариацияның тіндердегі транскриптомдағы вариацияны қалыптастырудағы рөлін түсінуге бағытталған адам генетикасы жобасы. Жоба өлгеннен кейінгі 700-ден астам донордан әртүрлі мата үлгілерін (> 50 түрлі тіндерді) жинады. Нәтижесінде> 11000 үлгі жиналды. GTEx тіндердің бөлінуін және тіндердің ерекшелігін түсінуге көмектесті EQTL.[26]
Сондай-ақ қараңыз
Әдебиеттер тізімі
- ^ Graur D, Zheng Y, Price N, Azevedo RB, Zufall RA, Elhaik E (20 ақпан 2013). «Теледидардың өлмейтіндігі туралы:» энкод «эволюциясы жоқ ізгі хабарға сәйкес адам геномындағы» функция «. Геном биологиясы және эволюциясы. 5 (3): 578–90. дои:10.1093 / gbe / evt028. PMC 3622293. PMID 23431001.
- ^ Гибсон G, Muse SV. Геном туралы ғылым (3-ші басылым). Сандерленд, MA: Sinauer Associates.
- ^ Pevsner J (2009). Биоинформатика және функционалды геномика (2-ші басылым). Хобокен, NJ: Уили-Блэквелл.
- ^ Ванг Х, Мэйхью Д, Чен Х, Джонстон М, Митра РД (мамыр 2011). «Қоңырау шалу карталары ДНҚ-мен байланысатын ақуыздардың геномдық мақсаттарын мультиплекстеп анықтауға мүмкіндік береді». Геномды зерттеу. 21 (5): 748–55. дои:10.1101 / гр.114850.110. PMC 3083092. PMID 21471402.
- ^ Хрдликова Р, Толоу М, Тянь Б (қаңтар 2017). «Транскриптомды талдаудың РНҚ-Сек әдісі». Вилидің пәнаралық шолулары: РНҚ. 8 (1): e1364. дои:10.1002 / wrna.1364. PMC 5717752. PMID 27198714.
- ^ Квасниески JC, Fiore C, Чаудхари HG, Коэн BA (қазан 2014). «ENCODE сегменттеу болжамдарының жоғары өнімді функционалды тестілеуі». Геномды зерттеу. 24 (10): 1595–602. дои:10.1101 / гр.173518.114. PMC 4199366. PMID 25035418.
- ^ Патвархан Р.П., Хиатт Дж.Б., Виттен Д.М., Ким МДж., Смит Р.П., мамыр D және т.б. (Ақпан 2012). «In vivo сүтқоректілердің күшейткіштерін жаппай параллельді функционалды диссекциялау». Табиғи биотехнология. 30 (3): 265–70. дои:10.1038 / nbt.2136. PMC 3402344. PMID 22371081.
- ^ Arnold CD, Gerlach D, Stelzer C, Boryń ŁM, Rath M, Stark A (наурыз 2013). «STARR-seq анықтаған геномдық сандық күшейткіш белсенділік карталары». Ғылым. 339 (6123): 1074–7. Бибкод:2013Sci ... 339.1074A. дои:10.1126 / ғылым.1232542. PMID 23328393. S2CID 54488955.
- ^ Өрістер S, Ән О (шілде 1989). «Ақуыз бен ақуыздың өзара әрекеттесуін анықтайтын жаңа генетикалық жүйе». Табиғат. 340 (6230): 245–6. Бибкод:1989 ж.340..245F. дои:10.1038 / 340245a0. PMID 2547163. S2CID 4320733.
- ^ Тиан С, Мунеруддин К, Чой МЖ, Тао Л, Бхуян РХ, Охми Ю, Фурукава К, Фурукава К, Боланд С, Шаффер С.А., Адам РМ, Донг М (27 қараша 2018). «Шига токсиндері мен рицинге арналған жалпы геномдық CRISPR экрандары гликозилдену үшін маңызды Гольджи ақуыздарын анықтайды». PLOS биологиясы. 16 (11). e2006951. дои:10.1371 / journal.pbio.2006951. PMC 6258472. PMID 30481169.
- ^ Харт Т, Чандрашехар М, Ареггер М, Штейнхарт З, Браун КР, МакЛеод Г және т.б. (Желтоқсан 2015). «Жоғары ажыратымдылықтағы CRISPR экрандары фитнес гендерін және генотипке тән қатерлі ісік аурулары бойынша жауапкершілікті анықтайды». Ұяшық. 163 (6): 1515–26. дои:10.1016 / j.cell.2015.11.015. PMID 26627737.
- ^ Shalem O, Sanjana NE, Hartenian E, Shi X, Scott DA, Mikkelson T, et al. (Қаңтар 2014). «Адам клеткаларындағы геномды CRISPR-Cas9 нокаут скринингі». Ғылым. 343 (6166): 84–87. Бибкод:2014Sci ... 343 ... 84S. дои:10.1126 / ғылым.1247005. PMC 4089965. PMID 24336571.
- ^ Гилберт Л.А., Хорлбек М.А., Адамсон Б, Виллалта Дж.Е., Чен Ю, Уайтхед Е.Х. және т.б. (Қазан 2014). «Геномдық масштабтағы CRISPR-гендік репрессия мен активацияны бақылау». Ұяшық. 159 (3): 647–61. дои:10.1016 / j.cell.2014.09.029. PMC 4253859. PMID 25307932.
- ^ Хорлбек М.А., Гилберт Л.А., Виллалта Дж.Е., Адамсон Б, Пак Р.А., Чен Ю, және т.б. (Қыркүйек 2016). «CRISPR арқылы генді репрессиялау және активтендіру үшін жаңа ұрпақтың ықшам және жоғары белсенді кітапханалары». eLife. 5. дои:10.7554 / eLife.19760. PMC 5094855. PMID 27661255.
- ^ Гасперини, Молли; Финдлей, Григорий М .; МакКенна, Аарон; Милбанк, Дженнифер Х .; Ли, Чоли; Чжан, Мелисса Д .; Кузанович, Даррен А .; Шендуре, Джей (тамыз 2017). «Мыңдаған үлкен, бағдарламаланған геномдық жою арқылы HPRT1 экспрессиясына қажет нормативтік элементтерді CRISPR / Cas9-делдалдық сканерлеу». Американдық генетика журналы. 101 (2): 192–205. дои:10.1016 / j.ajhg.2017.06.010. PMC 5544381. PMID 28712454.
- ^ Subramanian A, Tamayo P, Mootha VK, Mukherjee S, Ebert BL, Gillette MA және т.б. (Қазан 2005). «Гендер жиынтығын байытуды талдау: геномдық экспрессия профилдерін түсіндірудің білімге негізделген тәсілі». Америка Құрама Штаттарының Ұлттық Ғылым Академиясының еңбектері. 102 (43): 15545–50. Бибкод:2005PNAS..10215545S. дои:10.1073 / pnas.0506580102. PMC 1239896. PMID 16199517.
- ^ «Тапқырлық жүйелері». Архивтелген түпнұсқа 1999-01-25. Алынған 2007-12-31.
- ^ «Ariadne Genomics: Pathway Studio». Архивтелген түпнұсқа 2007-12-30 жж. Алынған 2007-12-31.
- ^ Винаягам А, Ху Ю, Кулкарни М, Ризель С, Сопко Р, Мохр SE, Перримон N (ақпан 2013). «Өткізгіштігі жоғары мәліметтер жиынтығы үшін ақуызды кешенді талдау жүйесі». Ғылыми сигнал беру. 6 (264): rs5. дои:10.1126 / scisignal.2003629. PMC 3756668. PMID 23443684.
- ^ Хилтон С.К., Дуд М.Б, Блум ДжД (2017). «фидмдер: терең мутациялық сканерлеу арқылы алынған филогенетикалық талдауға арналған бағдарламалық жасақтама». PeerJ. 5: e3657. дои:10.7717 / peerj.3657. PMC 5541924. PMID 28785526.
- ^ Diss G, Lehner B (сәуір 2018). «Физикалық өзара әрекеттесудің генетикалық ландшафтысы». eLife. 7. дои:10.7554 / eLife.32472. PMC 5896888. PMID 29638215.
- ^ Шмиедель, Йорн М .; Лехнер, Бен (17 маусым 2019). «Терең мутагенезді қолдану арқылы ақуыз құрылымын анықтау». Табиғат генетикасы. 51 (7): 1177–1186. дои:10.1038 / s41588-019-0431-x. PMID 31209395.
- ^ Роллинз, Натан Дж.; Брок, Келли П.; Пулвейк, Фрэнк Дж .; Стифлер, Майкл А .; Готье, Николас П .; Сандер, Крис; Маркс, Дебора С. (17 маусым 2019). «Мутациялық терең сканерлеу кезінде ақуыздың 3D құрылымын шығару». Табиғат генетикасы. 51 (7): 1170–1176. дои:10.1038 / s41588-019-0432-9. PMC 7295002. PMID 31209393.
- ^ Ганди М, Ли Д, Мохаммад-Нури М, Сыра MA (шілде 2014). «K-mer функцияларын қолдана отырып, реттелетін реттілікті жақсарту». PLOS есептеу биологиясы. 10 (7): e1003711. Бибкод:2014PLSCB..10E3711G. дои:10.1371 / journal.pcbi.1003711. PMC 4102394. PMID 25033408.
- ^ Li Y, Shi W, Wasserman WW (мамыр 2018). «Терең оқытудың бақыланатын әдістерін қолдана отырып, сис-реттеуші аймақтарды геномды болжау». BMC Биоинформатика. 19 (1): 202. дои:10.1186 / s12859-018-2187-1. PMC 5984344. PMID 29855387.
- ^ GTEx консорциумы; Зертхана, деректерді талдау және үйлестіру орталығы (Ldacc) - Талдау жұмыс тобы .; Статистикалық әдістер тобы - талдау тобы; GTEx (eGTEx) топтарын күшейту; NIH ортақ қоры; NIH / NCI; NIH / NHGRI; NIH / NIMH; NIH / NIDA; Биоспецименттерді жинаудың көзі - NDRI; Биоспецименттерді жинау көзі - RPCI; Биоспейсеннің негізгі ресурсы - VARI; Brain Bank репозиторийі - Майами университеті Brain Endowment Bank; Leidos Biomedical — Жобаларды басқару; ELSI Study; Геном шолғышының деректерін интеграциялау және визуалдау - EBI; Геном браузерінің деректерін интеграциялау және визуалдау - Ucsc Genomics Institute, Калифорния университеті Санта-Круз .; Жетекші талдаушылар; Зертхана, деректерді талдау және үйлестіру орталығы (Ldacc):.; NIH бағдарламасын басқару; Биоспекцияны жинау; Патология; eQTL қолжазба бойынша жұмыс тобы; Баттл, А .; Браун, С .; Энгельхардт, Б. Montgomery, S. B. (12 қазан 2017). «Адамның тіндеріндегі гендердің экспрессиясына генетикалық әсерлер» (PDF). Табиғат. 550 (7675): 204–213. Бибкод:2017 ж. 550..204А. дои:10.1038 / табиғат24277. PMC 5776756. PMID 29022597.
Сыртқы сілтемелер
- Функционалды геномика: OnLine поезында EBI ресурстарымен таныстыру
- Еуропалық ғылыми қордың функционалды геномика шекаралары бағдарламасы
- MUGEN NoE - Мутантты тышқан модельдеріндегі интеграцияланған функционалды геномика
- Табиғат туралы түсінік: функционалды геномика
- Биоинформатика және функционалды геномика - серіктес сайт Биоинформатика және функционалды геномика, 2-ші басылым.
- ҚОЙЫҢЫЗ
- Функционалды геномика және ауру бойынша 4-ші Еуропалық ғылыми қор конференциясы

